 |
 |
- Search
| Epilia: Epilepsy Commu > Volume 5(2); 2023 > Article |
|
Abstract
Background
Methods
Results
Conclusions
NOTES
Conflicts of interest
Ki-Young Jung is the Editor-in-Chief of Epilia and was not involved in the review process of this article. All authors have no other conflicts of interest to declare.
Author contributions
Conceptualization, Data curation, Formal analysis: YWS, KYJ. Funding acquisition, Project administration: KYJ. Investigation, Methodology: All authors. Visualization: YWS. Writing–Original Draft: YWS. Writing–Review & Editing: All authors.
All authors read and approved the final manuscript.
Fig. 1.
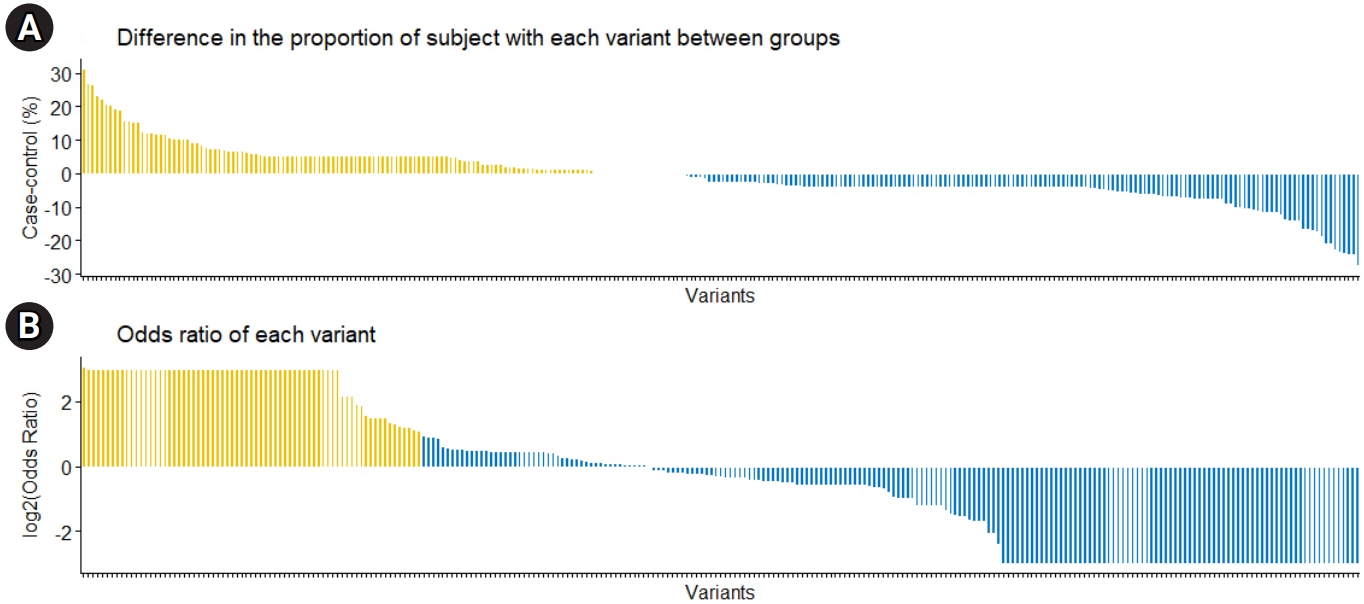
Table 1.
Table 2.
CHR, chromosome; REF, reference allele; ALT, altenative allele; RSID, reference single nucleotide polymorphism identification number of dbSNP (http://www.ncbi.nlm.nih.gov/snp/).
Table 3.
CHR, chromosome; REF, reference allele; ALT, altenative allele; RSID, reference single nucleotide polymorphism identification number of dbSNP (http://www.ncbi.nlm.nih.gov/snp/).
Table 4.
CHR, chromosome; REF, reference allele; ALT, altenative allele; RSID, reference single nucleotide polymorphism identification number of dbSNP (http://www.ncbi.nlm.nih.gov/snp/).






